Capacités et performances totales de l’offre de calcul
Total compute
27896
#CPU HT*
Total storage
15
#PB
Total RAM
45
#PB
Total GPU
44
#Card
* CPU HT = hyperthreadé
Choisir sa solution de calcul
L’ensemble de l’offre de calcul et de stockage de l’IFB est porté par le NNCR (Réseau national de ressources informatiques).
- Les plateformes participantes aux clusters : ABiMS, BiRD, GenoToul, GenOuest, IFB-BiGEst-Cluster, IFB Core, MicroScope, Migale, South Green
- Les plateformes participantes à la fédération du Cloud : AuBi, Bilille, BiRD, GenOuest, IFB-BigEst-Cloud, IFB-core, Prabi, CBP-PSMN
Ces fonctionnalités et capacités permettent de répondre à des usages et besoins d’analyse spécifiques.
Fonctionnalités et usages
Cluster
- Infrastructure de type HPC (High Performance Computer)
- Plusieurs interfaces d’accès : SSH, Galaxy et autres portails web
- Ressources bioinformatiques : générales et spécialisées
- Environnements logiciels via Conda et Singularity déjà configurés
- Solution adaptée aux utilisateurs biologistes et bioinformaticiens
- Niveaux d’expertise de l’utilisateur : du novice à l’expert
Cloud
- Offre à la demande des ressources de calcul et des données de référence
- Possibilité de déployer des appliances (machines préconfigurées) ou d’installer sa propre infrastructure bioinformatique.
- Grande flexibilité et contrôle total sur l’environnement
- Ressources à la demande
- Niveaux d’expertise de l’utilisateur : du novice à l’expert
Types d’analyses
Cluster
- Calculs intensifs nécessitant beaucoup de cœurs de calcul simultanément
- Analyses standardisées avec des outils déjà installés
- Travaux nécessitant un accès à de gros volumes de stockage partagé
Cloud
- Analyses bioinformatiques spécifiques nécessitant des environnements logiciels préconfigurés
- Projets nécessitant des configurations personnalisées
- Tests de nouveaux outils ou développements
Capacités techniques
Cluster
- 4300 cœurs (hyperthreadés)
- 2 Po de stockage
Cloud
- 6 000 cœurs de calcul
- 28 teraoctets (To) de mémoire
- Ressources réparties entre 7 sites
Localisation géographique
L’offre de calcul et de stockage de l’IFB est déployée au travers de notre réseau de plateformes membres.
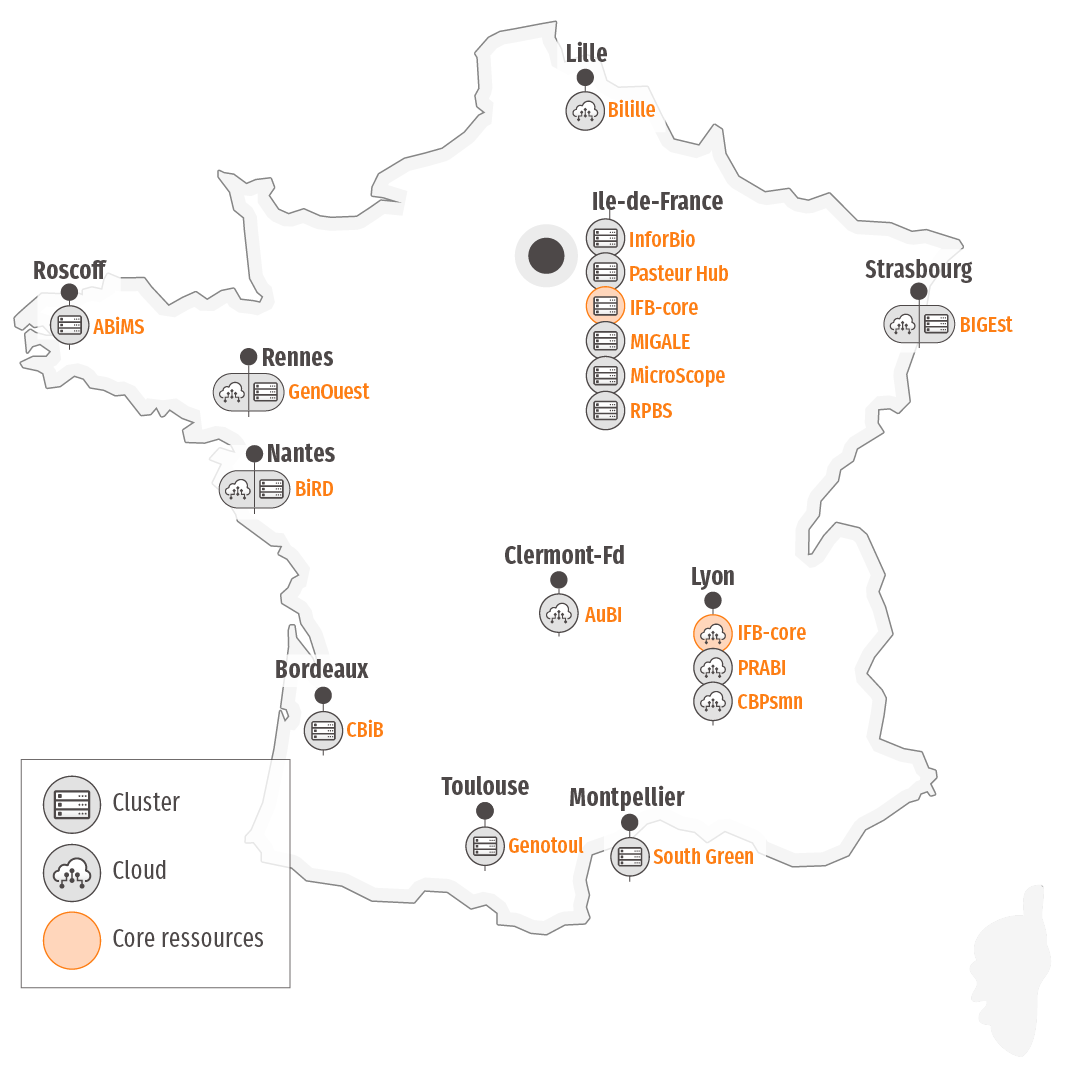

L’ensemble de l’offre de calcul et de stockage de l’IFB est porté par le NNCR (Réseau national de ressources informatiques).
Le Réseau national de ressources informatiques - NNCR
Les services aux communautés sont assurés par le NNCR, il comprend tout le matériel (IFB-core + plateformes régionales) contribuant à assurer l’offre de services. Le NNCR s’appuie sur une infrastructure distribuée constituée des 8 clouds et 6 clusters répartis sur 13 centres régionaux, et 2 ressources centralisées: les serveurs de l’IFB-core (cloud à Lyon, cluster à Orsay), et de neuf plateformes de calcul haute performance localisées dans différentes régions. Ce réseau est ouvert aux communautés de recherche française et internationale, publiques et privées, dans le domaine des sciences du vivant.
L’IFB joue également un rôle clé dans la stratégie nationale de mutualisation et de modernisation des infrastructures numériques scientifiques. Enfin, au niveau ELIXIR, ce service contribue à la maintenance de Biocontainers et à la coordination de la communauté Galaxy dans ELIXIR.
Par rapport au traitement des données dans d’autres domaines scientifiques, les caractéristiques particulières des données biologiques les rendent dépendantes d’une architecture matérielle, d’un environnement logiciel et de collections de données très spécifiques qui ne sont actuellement pas disponibles dans les centres de calcul nationaux. Pour faire face à ces défis, l’IFB a organisé ses services en décentralisant les ressources physiques, logistiques et humaines.
Une telle décentralisation des ressources offre au moins quatre avantages principaux :
> Le cloud IFB
La fédération de clouds propose un ensemble d’environnements préconfigurés, ce qui permet aux scientifiques et bioinformaticiens de choisir la configuration la mieux adaptée à leurs analyses. Ces environnements bioinformatique virtuels développés par certains membres de la communauté de l’IFB sont enregistrés dans le catalogue Biosphère. Les scientifiques et les ingénieurs peuvent lancer leurs propres environnements virtuels avec des ressources réservées qui peuvent être adaptées à leurs propres besoins sans interférer avec les autres utilisateurs. L’activité est en constante progression, et le cloud français de l’IFB a déjà été utilisé pour de nombreuses analyses scientifiques et des sessions de formation, des ateliers, des hackathons ou plusieurs sessions récurrentes d’écoles scientifiques ou d’universités.
Un compte offre un espace variable (de 25 Go à 3 To par machine virtuelle pour la durée de l’exécution des travaux mais supprimé à l’extinction de la machine virtuelle) auquel sera prochainement associé un espace de stockage persistant et partagé entre les machines virtuelles d’un utilisateur ou d’un groupe.
Vous pouvez aussi utiliser le cloud IFB comme support pour vos formations. Pour en savoir plus
> Les clusters en région
L’ensemble des clusters sur nos plateformes en région sont dédiés aux utilisateurs régionaux et thématiques.
> Le cluster IFB-core
Qu’est-ce que le cluster IFB-core ?
Le Core Cluster est l’infrastructure nationale de calcul de l’IFB. Il vise à répondre aux besoins de calcul de toutes les communautés dans les domaines de la santé et de la biologie, en mettant l’accent sur les utilisateurs qui ne disposent pas de ressources de calcul locales.
L’infrastructure est ouverte à tous les utilisateurs ayant une adresse e-mail académique en France ou dans l’un des pays membres d’ELIXIR.
Le Core Cluster est accessible via trois modalités :
L’administration du Core Cluster est réalisée en collaboration. Plus de six ingénieurs de cinq plateformes IFB construisent et contribuent au projet quotidiennement. Afin de gérer les multiples contributions, elles sont supervisés par des mécanismes CI (Ansible + Gitlab runner) connectés à un dépôt de code commun (Gitlab).
Documentation complète du Cluster : https://ifb-elixirfr.gitlab.io/cluster/doc/
Conditions d’utilisation : https://ifb-elixirfr.gitlab.io/cluster/doc/terms-of-usage/
Services
Gestion des comptes
Tout utilisateur·rice académique peut demander un compte pour le Core Cluster via notre portail de gestion et d’enregistrement des comptes : https://my.cluster.france-bioinformatique.fr. Attention, ce compte n’est valide que pour le Core Cluster et ne sera pas reconnu sur d’autres services de l’IFB.
Soumission de tâches informatiques
L’utilisation principale du cluster se fait avec SLURM à partir d’une console SSH.
Si vous êtes débutant·e avec SLURM, veuillez lire la documentation et le tutoriel du Core Cluster pour apprendre à soumettre vos premières tâches :
https://ifb-elixirfr.gitlab.io/cluster/doc/slurm/slurm_user_guide/
Portail web interactif
Le portail web Open Ondemand vous permet d’exécuter des outils interactifs comme RStudio ou Jupyterlab sur les ressources du cluster via une interface web simple. Trouvez la page de présentation, la documentation et la vidéo dédiée.
Galaxy
Le Cluster IFB Core fournit les ressources informatiques de l’instance française de Galaxy : usegalaxy.fr
usegalaxy.fr offre une large gamme d’outils de bioinformatique accessibles en ligne. Certains outils sont également accessibles via des sous-domaines thématiques tels que métabolomique, singleCell, covid19, etc.
Support technique
Le portail support.cluster.france-bioinformatique.fr permet aux utilisateur·rices du cluster de contacter notre équipe de support pour toute demande technique, y compris:
* Le support pour usegalaxy.fr est disponible sur le forum communautaire IFB
Soutien à la communauté de bioinformatique
Un forum communautaire vous permet d’échanger des idées avec des biologistes et des bioinformaticien·nes sur la bioinformatique : utilisations et options d’un outil, mise en place d’un flux de travail autour d’un thème spécifique, etc.
Hébergement de formations
Le Cluster IFB Core peut fournir des ressources informatiques pour votre session de formation. De nombreuses modalités sont proposées par notre équipe de support (création de comptes génériques temporaires, réservation de ressources du cluster, etc.).
Pour demander des ressources du cluster pour votre formation, remplissez le formulaire de demande.
Contribuez à la TaskForce du Cluster IFB-core
Si vous avez des compétences en outils bioinformatiques ou en administration système, rejoignez la TaskForce du Core Cluster IFB pour déployer vos outils ou contribuez à la gestion de l’infrastructure IFB et entrez dans la légende. N’hésitez pas à nous contacter en envoyant un e-mail à :
contact-nncr-cluster@groupes.france-bioinformatique.fr . Sinon, si vous souhaitez être formé aux technologies utilisées sur l’infrastructure, des sessions de formation/tutorat sont régulièrement proposées.
Intégration continue (CI) & Travail collaboratif
L’administration des ressources est réalisée de manière collaborative. Afin de gérer plusieurs contributions, celles-ci sont gérées par un mécanisme d’intégration continue connecté à un dépôt de code commun.
Traçabilité & Contributions sécurisées
Toutes les actions d’installation, de paramétrage et de maintenance doivent être traçables autant que possible. Cela afin de :
Nous avons choisi d’utiliser un dépôt Git hébergé sur GitLab. Git répond à tous nos besoins en matière de traçabilité. Quant à l’interface GitLab, elle nous fournit un espace pour échanger des informations et offre la possibilité de travailler avec des Merge Request (Pull Request) et d’héberger nos propres runners de jobs CI. Ces MR nécessitent l’insertion d’une phase de révision de code avant la mise en production. Chaque modification ou ajout est ainsi validé par une révision par les pairs afin d’éviter les erreurs et de s’assurer qu’au moins deux personnes en ont connaissance.
Membres de la TaskForce :