Pour cette nouvelle édition du BioHackathon Europe, organisé par ELIXIR Europe, les participants et la délégation ELIXIR France se sont rendus du 3 au 7 novembre 2025 à Berlin. Cet événement collaboratif international a rassemblé environ 175 (en présentiel) et 290 (en remote) chercheurs, bioinformaticiens et développeurs pour travailler sur des projets visant à relever des défis de recherche en Bioinformatique. Ce hackathon annuel est un lieu d’accélération de projets pour innover dans des domaines comme l’intégration des données, la science des données et l’application des principes FAIR (Findable, Accessible, Interoperable, Reusable), où les participants mettent en œuvre leurs idées lors de sessions de codage intensives.
Ce Biohackathon a permis et permet chaque année aux projets de générer de nouveaux développements (intégration de prototypes, propositions de preuve de concept ou ébauches à la production), de tester de nouvelles versions, recueillir des informations pour les nouvelles fonctionnalités, atteindre de nouveaux utilisateurs, ou encore recevoir des avis d’experts dans différents domaines…
Cette année, nos équipes ont déposé 4 projets parmi les 31 retenus au BioHackathon, marquant encore une belle implication des équipes du nœud français.
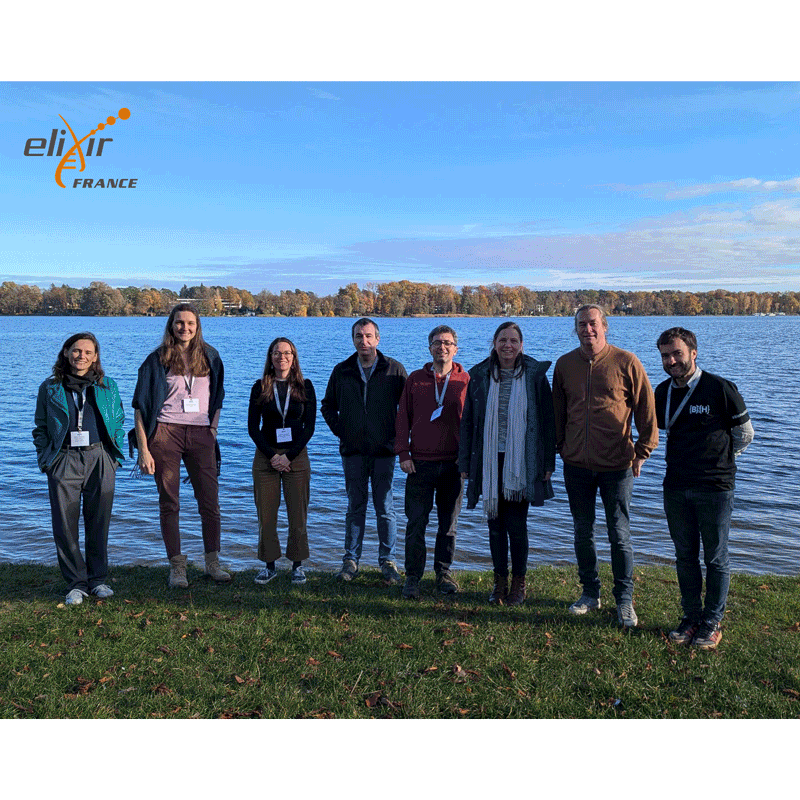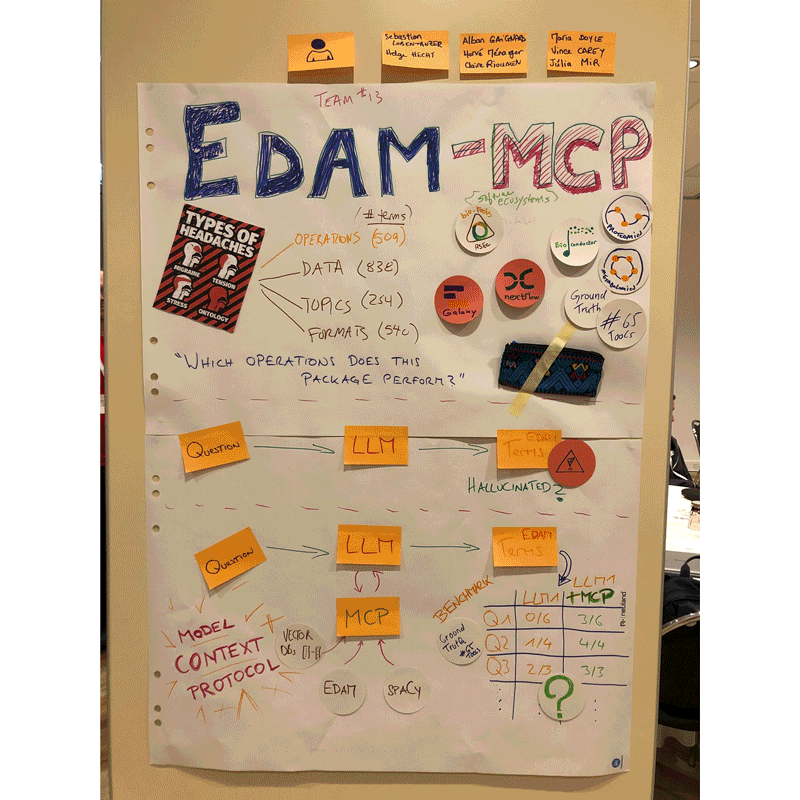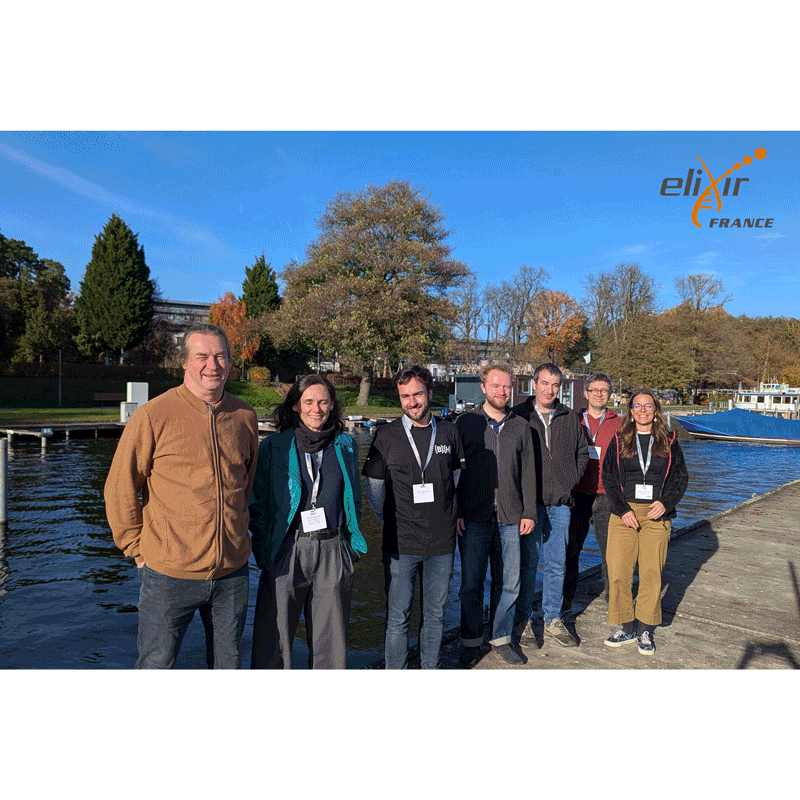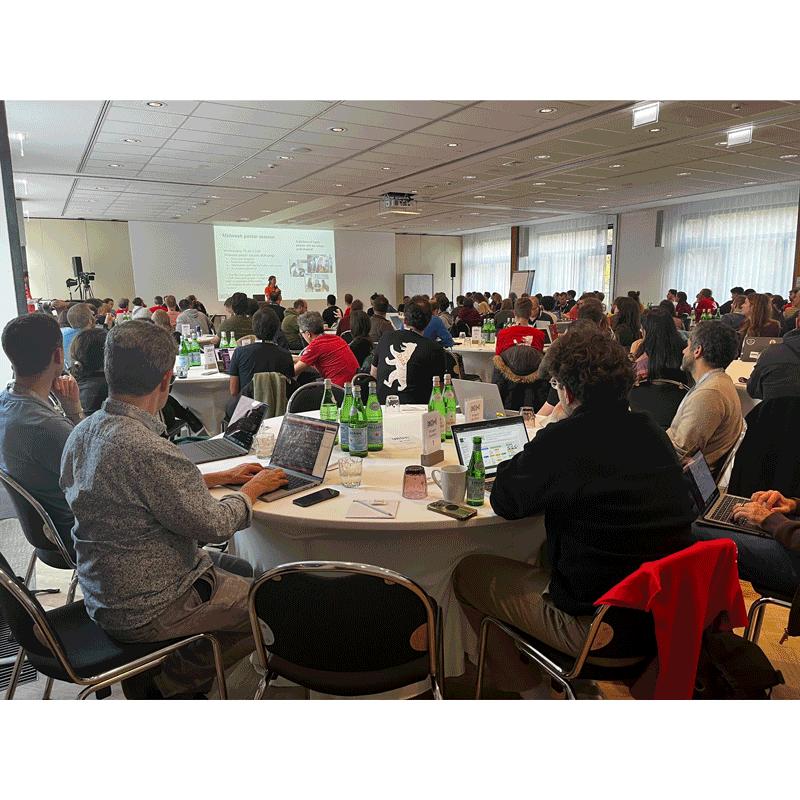
Projet 13 – Improving package annotation in metabolomics and proteomics via robust, ontology-driven LLM integration par Claire Rioualen, co-animé avec Sebastian Lobentanzer (ELIXIR-DE) et Helge Hecht (ELIXIR-CZ)
Notre projet s’est focalisé sur le développement d’EDAM MCP (Model Context Protocol), un outil pour faciliter l’annotation de ressources bioinformatiques avec l’ontologie EDAM. Afin de tester la validité du prototype, nous avons rassemblé un ensemble d’outils et annotations de référence dans le domaine de la métabolomique, montrant des résultats probants. Ce BioHackathon nous a permis de réunir sur place des collaborateurs avec des expertises variées, venant de toute l’Europe et des États-Unis.
Projet 14 – Metabolomics and Proteomics file format interoperability fest par Franck Giacomoni
Ce projet a rassemblé une quarantaine de participant·es autour du format mzTab-M, lors du BioHackathon ELIXIR Europe 2025 à Bad Saarow et dans un satellite à Clermont-ferrand. L’équipe a d’abord cartographié les implémentations logicielles de mzTab-M, collecté des jeux de données réelles de métabolomique et lipidomique et automatisé leur validation via le validateur jmzTab-M et des workflows GitHub dans le dépôt HUPO-PSI. Une campagne d’« interopérabilité croisée » a ensuite permis de tester une vingtaine de combinaisons entre outils producteurs (mzmine, MS-Dial, xcms, LipidDataAnalyzer, Progenesis QI, MetaboScape, MASSter) et plateformes consommatrices (GNPS, LipidCompass, MetFamily, MetaboLights, MetaboAnalyst), avec une majorité de conversions réussies. Ce travail a permis la correction de bugs, d’ouvrir des tickets structurés et de discuter de la consolidation de bibliothèques communes en Python et R et faciliter leur adoption et leur maintenance à long terme. Le projet a également renforcé l’intégration de mzTab-M dans des écosystèmes important pour la communauté comme Galaxy, PeakForest, EDAM et bio.tools. Enfin, un groupe dédié a esquissé les évolutions nécessaires de la spécification pour par exemple, mieux gérer les plans expérimentaux multifactoriels ou les ambiguïtés d’annotation des métabolites.
« Ce BioHackathon aura permis de faire progresser la communauté à la fois en rendant plus robuste les différentes implémentations du format mzTab-M mais également en amenant des discussions éclairées sur les évolutions futures nécessaire des spécifications de ce format, préparant un cadre très important pour une métabolomique et une lipidomique d’avantage reproductible, comparable et interopérable. »
Projet 16 – MiCoReCa (Microbiome Community Resource Catalogue): Towards centralised curation and integration par Bérénice Batut, co-animé avec Nikolaos Strepis (ELIXIR-NL) et Vivek Ashokan (ELIXIR-FR)
Notre projet a développé un processus de collecte, filtrage et curation des ressources ELIXIR dédiées au microbiome (outils, workflows, packages bioconda). Nous avons défini des mots-clés ciblés, extrait et initié la curation manuelle, et identifié des termes EDAM manquants. Nous collaborons maintenant avec la communauté ELIXIR Microbiome pour valider ces mots-clés, curer les ressources et proposer de nouveaux termes EDAM. Ce cadre méthodologique, conçu pour être réplicable, sera étendu aux communautés biodiversité et single-cell omics. Ce travail s’aligne avec les projets MUDIS4LS et Cloud4SAMS, ainsi qu’avec le Groupe de Travail « Catalogue des ressources » de la communauté Microbiologie (C4) de l’IFB.
Projet 21 – Opening the TMD: making it more Usable, Visible, and Connected par Olivier Sand, co-animé avec Nina Norgren (ELIXIR-SE) et Eleni Adamidi (ELIXIR-GR)
Notre projet avait pour but de développer une API et des widgets pour la base de données Training Metrics Database qui concentre les données démographiques, qualité et impact des formations des nœuds ELIXIR. L’API fonctionne pour récupérer les données agrégées sur l’ensemble des nœuds. Il reste à ajouter la partie permettant d’ajouter et récupérer les données d’un nœud en particulier. Des morceaux de codes paramétrables à intégrer dans les sites web sont disponibles pour visualiser les données agrégées sur l’ensemble des nœuds. La fonctionnalité permettant de le faire pour les données d’un seul nœud est en cours de développement. Le BioHackathon a également permis d’améliorer l’interface utilisateurs.
